algorithmic Me
COVID-19: Visualizando datos
En el repositorio COVID-19 comparto una serie de pequeños programas escritos en Python3 para visualizar la evolución de epidemia en las distintas partes del mundo. Están hechos para realizar distintos gráficos a partir de las siguientes bases de datos:
- 2019 Novel Coronavirus COVID-19 (2019-nCoV) Data Repository by Johns Hopkins CSSE.
- Reportes del Ministerio de Salud de la Nación Argentina.
- Datasets en datos.gob.ar.
los datos
Cargo manualmente los datos de los Informes diarios del Ministerio de Salud de la Nación
Argentina en una planilla de cálculos que después exporto en formato csv. Este archivo tiene un
montón de huecos ya que los informes no detallan por provincia algunos datos. Mientras la serie temporal de
casos confirmados y fallecimientos está completa con los datos de todas las provincias del país,
los datos de altas, casos descartados y pruebas de laboratorio están incompletos. En todas
las categorías existe un apartado UNKNOWN para asegurar que el total coincida con los informes
consultados.
Para visualizar datos de 2019 Novel Coronavirus COVID-19
(2019-nCoV) Data Repository by Johns Hopkins CSSE o los
Datasets in datos.gob.ar hace falta descargar los archivos correspondientes. Cuando los datos cargados en
Argentina.csv son procesados se crean un conjunto de archivos csv que almacenan distinta
información. Por ejemplo: casos diario, fallecimientos diarios o casos diarios
tomando el promedio de los últimos 3 o 5 días. Los gráficos de la situación argentina toman datos de estos
archivos.
los gráficos
Pueden obtenerse distintos gráficos para visualizar la información por fecha o también alineando las series a partir de que se cumple una condición (un cierto número de casos confirmados o fallecimientos). Todos los programas tienen una lista de variables en las que se puede decidir qué gráficos se muestran al correr el código. Guardo los que me parecen significativos en la carpeta Argentina_Data/actual_charts.
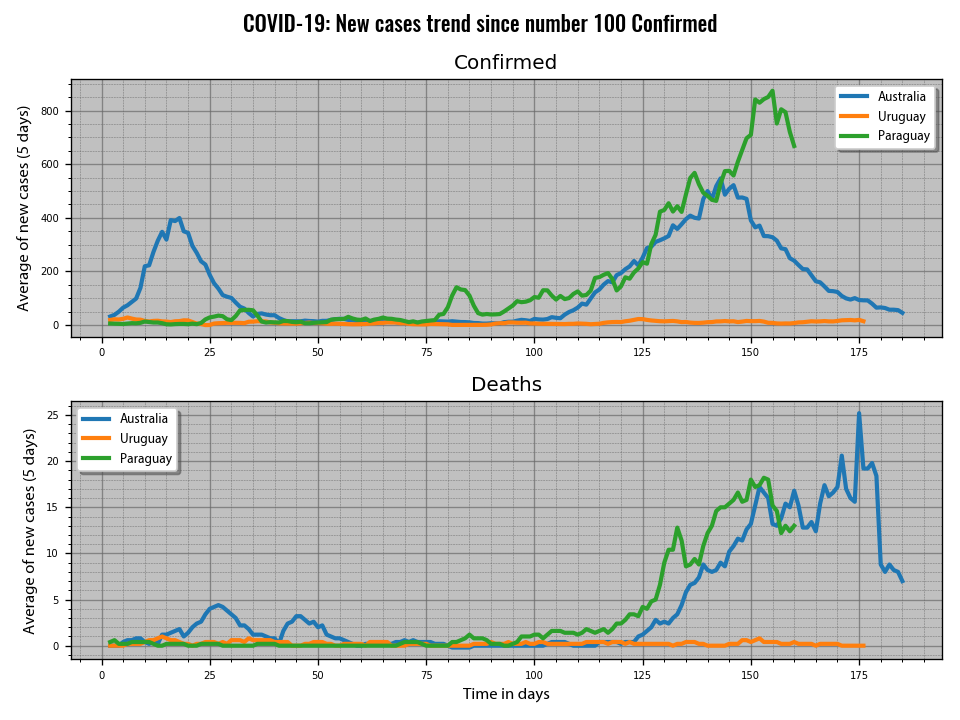
las definiciones
Los gráficos y los archivos csv construidos a partir de los datos de la situación del brote en Argentina contienen varias categorías. Entender a qué se refiere cada una es fundamental para interpretar los datos correctamente. Por eso hay que tener en cuenta las siguientes definiciones:
- Caso confirmado: representa a una persona infectada detectada por las autoridades sanitarias.
- Casos confirmados: el número total de casos confirmados hasta el momento.
- Casos activos: los casos confirmados menos las altas médicas (confirmados - recuperados).
- Fallecimientos: el número de personas fallecidas a causa de la enfermedad confirmado por las autoridades.
- Casos/fallecimientos diarios: el número de nuevos casos o fallecimientos registrados en un día. Se obtienen restando los casos de dos días sucesivos (cn+1 - cn). También se guardan tendencias tomando promedios para 3 y 7 días.
- Tasa de mortalidad: la proporción de casos confirmados que fallece (fallecidos / (casos confirmados)).
- Tiempo de duplicación (lineal): la cantidad de días necesarios para que los casos acumulados se dupliquen si los casos diarios se mantuvieran constantes. También se calculan tomando promedios para 3 y 5 días. Es importante tener en cuenta que no es la forma más frecuente de calcular el tiempo de duplicación (que asume un crecimiento exponencial).
- Tasa de positividad: la proporción de pruebas de laboratorio que dan un resultado positivo (positivos / (pruebas de laboratorio)). Se calcula para las pruebas diarias, tomando promedios para 3 días y de manera acumulativa.
- Estimación a partir de fallecimientos: una estimación de la cantidad de infectados real a partir del número de fallecimientos teniendo en cuenta una cierta tasa de mortalidad real y el tiempo promedio que tarda un paciente en morir a partir de la aparición de los síntomas.
- Tasa de detección: la proporción de los casos reales estimados detectados por las autoridades (casos confirmados).